この記事は cythonを使ってMCMCを実装 | サンプリング編 の続きです.
前回の記事でoutputされたデータを図示する.
コードは,git clone https://github.com/akitoshiblog/blogsiteでmcmc_cythonの下にある.
サンプリングの確認
Rosenbrock’s banana functionに対して,百万回サンプルして,実際データとして使うのは千回に一個とした.
始めに以下のmoduleをimportする.図示のコードが書かれているのは,tracer.pyである.こちらはクラスで書かれているので,手に入れたデータを更に加工して使う場合は,クラスを継承させて処理させればよい.
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
%matplotlib inline
import seaborn as sns
sns.set(context="paper" , style ="whitegrid",rc={"figure.facecolor":"white"})
# custom function
import tracer
以下のメソッドを用いると次のような出力になる.デフォルトではmeanとmedian, HPD95%の計算の際には,最初の10%のデータを捨てるようしている.
path = "output/result_20200119_215855.txt"
trace = tracer.tracer(path,{"samp":0.2,"alpha":0.05})
trace.getData()
print(trace.comment)
trace.printSummary(m="mean")
trace.printSummary(m="median")
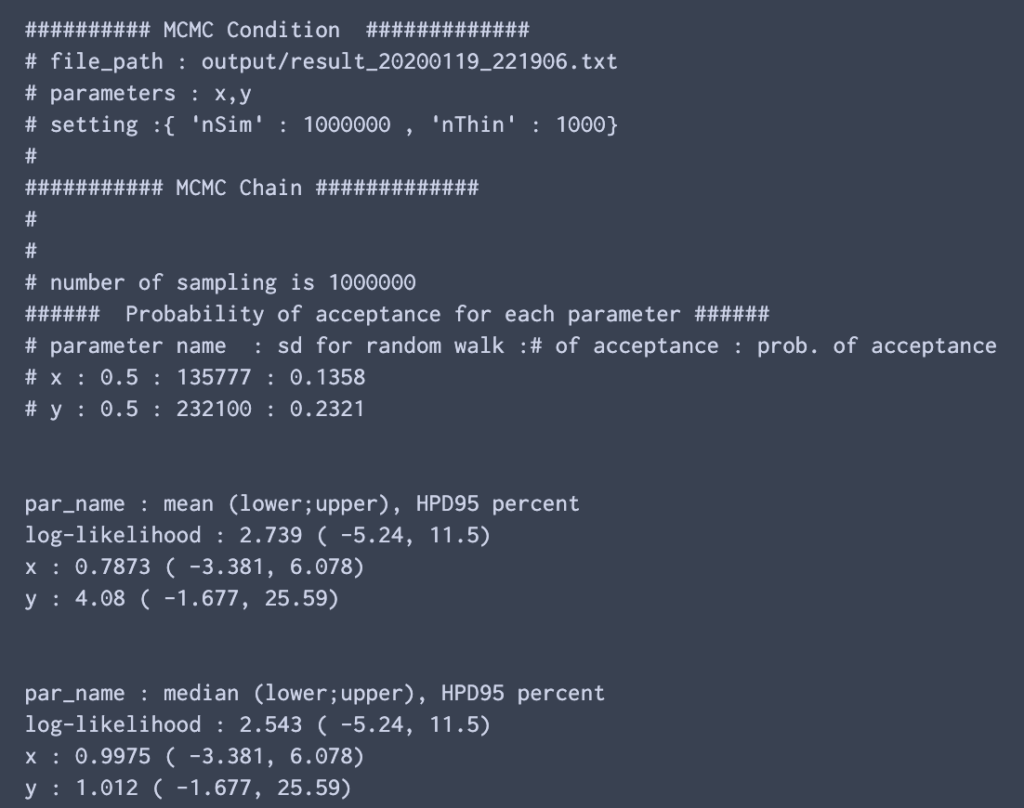
以下のコードで,log-likelihoodと全てのパラメータ,time (sec/M) に関して,sampleに対する値とヒストグラムを表示する.
trace.plotParameters()
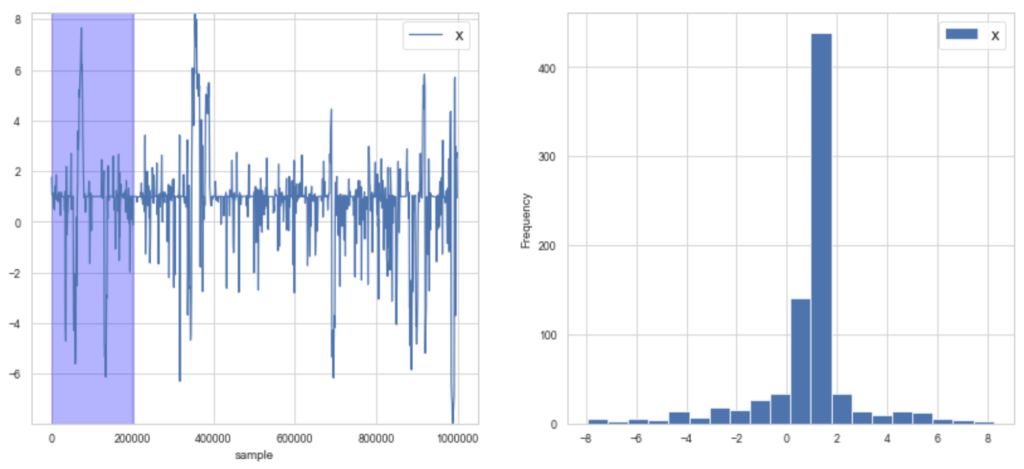
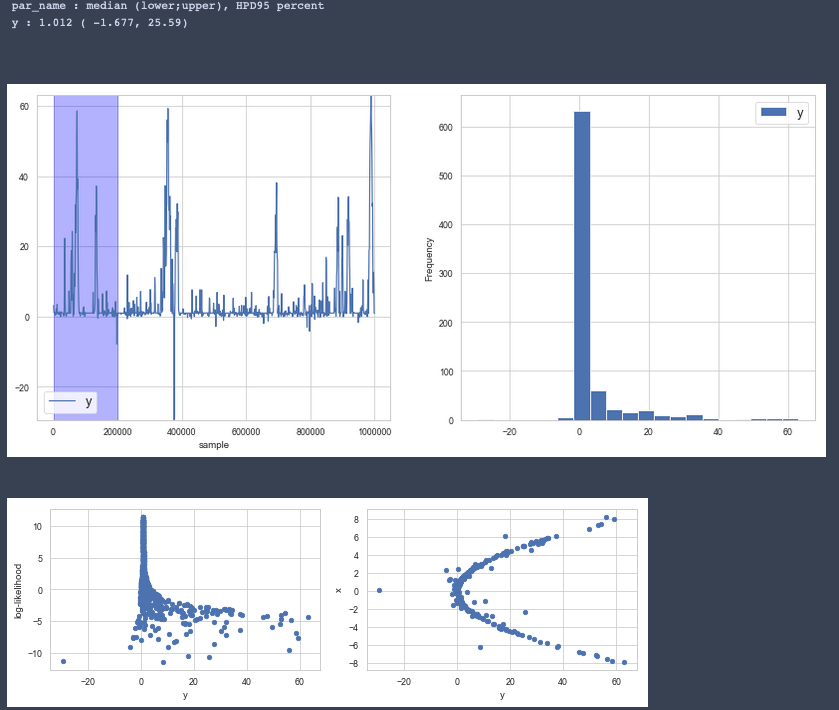
このコードで,あるパラメータに関しての図を表示する.上の二つに加えて,各パラメータとの相関図も作成する.
trace.checkOneParam("y")
———-雑感(`・ω・´)———-
結果については,meadianなら大域解に収束しているが,平均だと違う値になってしまっている.ランダムウォーク用のsdの設定が自分で調整しなければいけなくなっているのでそこは改善の余地あり…


コメント
[…] メインのコードは.myMCMC.pyx/myMCMC.pxdとmyStats.pxd/myStast.pyxである.図示に関しては cythonを使ってMCMCを実装 | 図示編 を参照のこと. […]